Programmierung Neuronaler Netze mit Keras
Bisher haben wir eine eigene Implementierung Neuronaler Netze angegeben, die helfen soll, deren grundlegende Funktionsweise zu vermitteln. Für die Praxis ist unsere Implementierung nicht geeignet. Keras ist eine Programmier-Schnittstelle, mit deren Hilfe sich Algorithmen zum Maschinellen Lernen inlusive Neuronaler Netze auf einer vielseitigen und effizienten Basis (wie TensorFlow) implementieren lassen. Im folgenden wollen wir Keras anhand von Beispiel-Programmen kennenlernen.
Logische XOR-Verknüpfung
Wir haben bereits gesehen, dass ein einzelnes Neuron nicht ausreicht, um die XOR-Verknüpfung auf zwei Eingängen zu lernen. Mit mehreren Neuronen ist das aber möglich. Bevor wir komplexere Funktionen lernen, implementieren wir dieses Beispiel in Keras.
Die folgenden Definitionen speichern wir in einer Datei xor.py. Die ersten Zeilen importieren benötigte Bibliotheken.
import numpy
import tensorflow.keras
import matplotlib.pyplot
Nun definieren wir die Struktur unseres Neuronalen Netzes. Wir verwenden zwei Neronen in der ersten Schicht und ein weiteres in der zweiten Schicht. In Keras können wir Neuronale Netze mit mehreren Schichten mit Hilfe von tensorflow.keras.models.Sequential definieren. tensorflow.keras.layers.Dense liefert eine Schicht, in der alle Neuronen mit allen vorherigen Neuronen verbunden sind. Wir können Schichten mit der Methode add einem Neuronalen Netzwerk hinzufügen.
network = tensorflow.keras.models.Sequential()
network.add(tensorflow.keras.layers.Dense(2, input_dim=2, activation="sigmoid"))
network.add(tensorflow.keras.layers.Dense(1, activation="sigmoid"))
Der ersten Schicht übergeben wir die Anzahl der Eingaben als zusätzlichen Parameter input_dim. Jede Schicht kann zudem eine unterschiedliche Aktivierungsfunktion übergeben bekommen, die für Neuronen dieser Schicht verwendet wird. Wir verwenden die Sigmoidfunktion. Ohne weitere Parameter werden die Neuronen standardmäßig mit zufälligen Gewichten initialisiert.
Bevor wir das erzeugte Netz trainieren können, müssen wir es mit der Methode compile darauf vorbereiten.
network.compile(loss="mean_squared_error", optimizer="sgd", metrics=["acc"])
Als Parameter übergeben wir den Namen einer Fehlerfunktion (hier wie zuvor basierend auf Fehlerquadraten) und einen Trainings-Algorithmus. Der Name sgd steht hier für stochastic gradient descent also das zuvor diskutierte Gradienten-Abstiegs-Verfahren. Der Parameter metrics erlaubt es, zusätzliche Metriken anzugeben, die während des Trainings berechnet werden sollen. Die Abkürzung acc steht hier für accuracy, den Anteil der Trainings-Beispiele für die das Netz das richtige Ergebnis liefert.
Training
Die Trainingsdaten erwartet Keras in Form von NumPy-Arrays. NumPy ist eine Numerik-Bibliothek für Python.
inputs = numpy.array([[0, 0], [0, 1], [1, 0], [1, 1]])
targets = numpy.array([0, 1, 1, 0])
Als Eingaben deklarieren wir ein mehrdimensionales Array inputs mit vier Einträgen von denen jeder zwei Eingaben der XOR-Funktion beschreibt. Das Array targets beschreibt die zu den Eingaben gehörigen erwarteten Ausgaben.
Nun können wir unser Netzwerk mit der fit-Methode trainieren.
training = network.fit(inputs, targets, epochs=20000, batch_size=1)
Der Parameter epochs beschreibt, wie oft die intern gespeicherten Gewichte aktualisiert werden sollen; batch_size gibt an, wieviele (zufällig ausgewählte) Trainings-Beispiele für eine solche Aktualisierung herangezogen werden.
Wir können nun das erstellte Programm im Terminal ausführen.
# python3 xor.py
...
4/4 [==============================] - 0s 746us/step - loss: 0.2493 - acc: 0.5000
Epoch 9781/20000
...
Es gibt während des Trainings Informationen zum Trainingsfortschritt aus. Die Angabe acc: 0.5000 zeigt an, dass das Netz für 50% (also 2 von 4) der Trainings-Beispiele das richtige Ergebnis liefert.
Wir erweitern nun das Programm so, dass es den Trainingsfortschritt auch grafisch darstellt. Dazu greifen wir auf das Objekt training zu, das von der fit-Methode zurückgeliefert wurde.
fig = matplotlib.pyplot.figure()
pic = fig.add_subplot(1,1,1)
pic.plot(training.history['loss'])
pic.plot(training.history['acc'])
pic.set_title('XOR training')
pic.legend(['loss','acc'], loc='upper left')
pic.set_xlabel('epoch')
pic.set_ylabel('metric')
fig.savefig('xor_training.png')
Wenn wir das erweiterte Programm ausführen, erzeugt es nach dem Training die folgende Grafik.
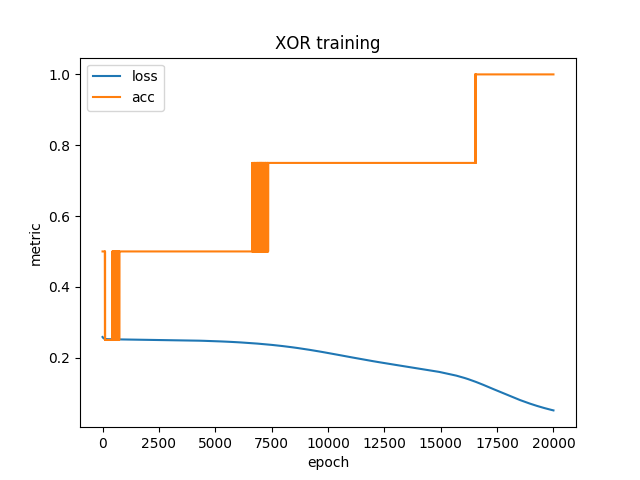
Der blau dargestellte Gesamtfehler (loss) nimmt kontinuierlich ab während der Anteil Trainingsbeispiele mit richtigem Ergebnis (acc) sprunghaft zunimmt und teilweise fluktuiert.
Auffällig ist, dass das Training sehr viele Schritte benötigt. Da Gewichte zufällig initialisiert und Trainingsbeispiele zufällig ausgewählt werden, führt jeder Programmlauf zu einem anderen Ergebnis. Nicht immer ist der Anteil korrekt klassifizierter Trainingsbeispiele am Ende bei 100%.
Um das Training zu beschleunigen können wir die Lernrate anpassen. Sie beschreibt, wie stark Gewichte in jedem Schritt angepasst werden. Die Standard-Implementierung des Gradienten-Abstiegs-Verfahrens verwendet eine Lernrate von 0.01. Um sie anzuheben, passen wir den Aufruf der compile-Methode wie folgt an.
sgd = tensorflow.keras.optimizers.SGD(learning_rate=0.5)
network.compile(loss="mean_squared_error", optimizer=sgd, metrics=["acc"])
Nun genügen oft schon 500 Schritte (statt den vorherigen 20000), um alle Testbeispiele korrekt zu klassifizieren, wie die folgende Grafik zeigt.
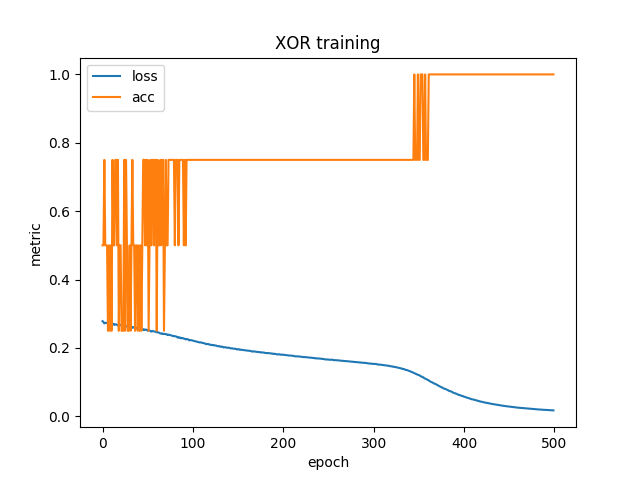
Bei zu hoher Lernrate kann es jedoch passieren, dass die Gewichte in jedem Schritt so stark verändert werden, dass kein Minimum für die Fehlerfunktion mehr gefunden wird.
Exkurs: NumPy-Arrays
Bevor wir ein komplexeres Beispiel behandeln, sehen wir uns NumPy-Arrays etwas genauer an. Wir haben bereits gesehen, dass wir Arrays mit der Funktion numpy.array erzeugen können.
>>> data = numpy.array([[0,0,0],[0,1,1],[1,0,1],[1,1,0]])
>>> print(data)
[[0 0 0]
[0 1 1]
[1 0 1]
[1 1 0]]
Darüber hinaus benötigen wir Methoden, die es erlauben, Teil-Arrays zu extrahieren. Dazu können wir zu extrahierende Bereiche mit einem Doppelpunkt zwischen eckigen Klammern notieren:
>>> print(data[1:3])
[[0 1 1]
[1 0 1]]
Die erste Zahl ist der Index, bei dem der extrahierte Teil beginnt. Die zweite Zahl ist der erste nicht mehr extrahierte Index. Negative Indizes erlauben es, Elemente vom Ende her abzuzählen. Wenn man eine oder beide Zahlen weglässt wird vom Anfang und/oder bis zum Ende extrahiert:
>>> print(data[1:-1])
[[0 1 1]
[1 0 1]]
>>> print(data[:])
[[0 0 0]
[0 1 1]
[1 0 1]
[1 1 0]]
Wir können mehrere Bereiche hintereinander notieren, um innerhalb mehrdimensionaler Arrays zu extrahieren. Der folgende Aufruf extrahiert alle Einträge des Arrays data, von jedem aber nur die ersten beiden Elemente (alle bis auf das letzte).
>>> print(data[:,:-1])
[[0 0]
[0 1]
[1 0]
[1 1]]
Dieses Array haben wir als Eingaben im Training der XOR-Funktion verwendet. Der folgende Aufruf extrahiert aus dem Array data die von uns für das Training der XOR-Funktion verwendeten erwarteten Ausgaben.
>>> print(data[:,-1])
[0 1 1 0]
Statt eines Bereiches können wir also auch einen einzigen Index (ohne Doppelpunkt) angeben.
Praxisbeispiel
Wir wollen nun ein Neuronales Netz trainieren, auf Basis gewisser Patientendaten vorherzusagen, ob bei zugehörigen Patienten innerhalb von fünf Jahren Diabetes ausgebrochen ist. Die CSV-Datei mit Patientendaten enthält neun Spalten. Was die ersten acht Spalten bedeuten, können Sie in der Beschreibung nachlesen. Die letzte Spalte gibt an, ob beim zugehörigen Patient innerhalb von fünf Jahren Diabetes ausgebrochen ist. Diese Spalte soll unser Neuronales Netz also auf Basis der ersten acht Spalten vorherzusagen lernen.
Zunächst lesen wir dazu die Daten aus der CSV-Datei in ein NumPy-Array ein.
import numpy
import tensorflow.keras
import matplotlib.pyplot
data = numpy.loadtxt('diabetes.csv', delimiter=',')
inputs = data[:,:-1]
targets = data[:,-1]
Nun erzeugen wir ein Neuronales Netz mit drei Schichten. Die erste Schicht hat zwölf Neuronen, die zweite acht und die letzte eines zur Ausgabe einer Null oder Eins.
network = tensorflow.keras.models.Sequential()
network.add(tensorflow.keras.layers.Dense(12, input_dim=8, activation='sigmoid'))
network.add(tensorflow.keras.layers.Dense(8, activation='sigmoid'))
network.add(tensorflow.keras.layers.Dense(1, activation='sigmoid'))
network.compile(loss='mean_squared_error', optimizer='sgd', metrics=['acc'])
Anders als bisher teilen wir die Datensätze nun in zwei Hälften. Die eine Hälfte wird für das Training verwendet wie bisher. Die andere Hälfte dient zur Kontrolle (validation) des trainierten Netzes und hat selbst keinen Einfluss auf die Gewichte. Über den Parameter validation_split teilen wir der fit-Methode mit, welcher (zufällige) Anteil der Daten für die Kontrolle verwendet werden soll.
training = network.fit(
inputs, targets, epochs=2000, batch_size=10,
validation_split=0.5
)
Diesmal verwenden wir in jedem Schritt zehn Trainingsbeispiele, um eine gemittelte Gewichts-Änderung zu bestimmen und führen insgesamt 2000 Anpassungen durch.
# python3 diabetes.py
...
Epoch 2000/2000
384/384 [==============================] - 0s 115us/step - loss: 0.1853 - acc: 0.7135 - val_loss: 0.2047 - val_acc: 0.6406
Wir können erkennen, dass bei den Kontrolldaten der Fehler größer und der Anteil korrekt klassifizierter Beispiele kleiner ist. Den Trainingsfortschritt stellen wir auch wieder grafisch dar. Diesmal erzeugen wir getrennte Kurven für die zum Training und die zur Kontrolle verwendeten Daten.
fig = matplotlib.pyplot.figure()
pic = fig.add_subplot(1,1,1)
pic.plot(training.history['loss'])
pic.plot(training.history['acc'])
pic.plot(training.history['val_loss'])
pic.plot(training.history['val_acc'])
pic.set_title('diabetes training')
pic.legend(['loss','acc','val_loss','val_acc'], loc='upper left')
pic.set_xlabel('epoch')
pic.set_ylabel('metric')
fig.savefig('diabetes_training.png')
Das erweiterte Programm erzeugt die folgende Grafik.
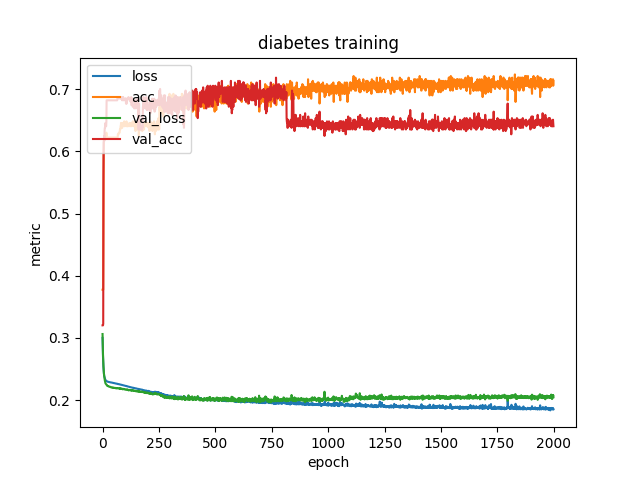
Wir können erkennen, dass der Anteil korrekt klassifizierter Kontrolldaten zwischen 750 und 1000 Schritten deutlich abnimmt, nachdem er vorher gemeinsam mit dem entsprechenden Anteil der Trianingsdaten zugenommen hatte. Diesen Effekt nennt man Überanpassung (overfitting). Das Neuronale Netz lernt dabei spezifische Eigenschaften der Trainingsdaten, die bei einer Klassifizierung anderer Daten nicht helfen sondern stören.
Trainings-Abbruch
Wir wollen nun das Programm so erweitern, dass das Training abgebrochen wird, sobald es Anzeichen für eine Überanpassung gibt. Statt des Anteils korrekt klassifizierter Kontrolldaten betrachten wir hierzu den zugehörigen Gesamtfehler, weil dieser weniger stark fluktuiert. Wir erweitern den Aufruf der fit-Methode um einen sogenannten Callback, der das Training abbricht, wenn sich der Gesamtfehler bei der Klassifizierung aller Kontrolldaten (val_loss für validation-set loss) 500 Schritte lang nicht mehr verringert.
stop = tensorflow.keras.callbacks.EarlyStopping(monitor="val_loss", patience=500)
training = network.fit(
inputs, targets, epochs=2000, batch_size=10,
validation_split=0.5, callbacks=[stop]
)
Das angepasste Programm bricht nach etwa 1300 Schritten ab und erzeugt die folgende Grafik.
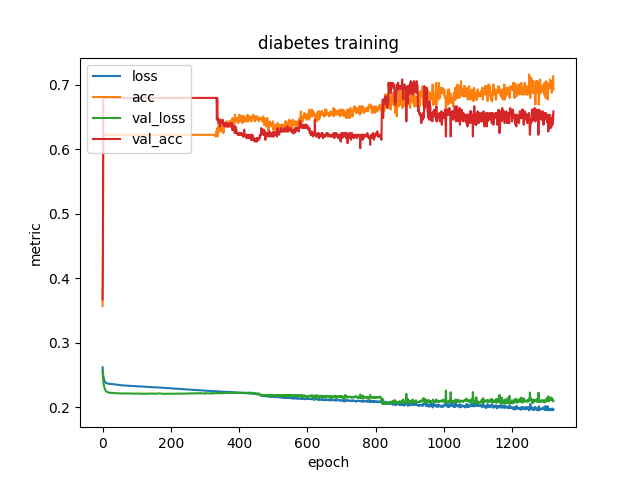
Da das letztlich erzeugte Netz noch immer (wenn auch nicht mehr so stark) an die Trainingsdaten überangepasst ist, erweitern wir das Training um einen weiteren Callback. Wannimmer der Gesamtfehler der Kontrolldaten ein neues Minimum erreicht, speichern wir das aktuell trainierte Netz in einer Datei best.h5.
best = tensorflow.keras.callbacks.ModelCheckpoint(
"best.h5", monitor="val_loss", save_best_only=True
)
training = network.fit(
inputs, targets, epochs=2000, batch_size=10,
validation_split=0.5, callbacks=[stop,best]
)
Das beim Training gespeicherte Neuronale Netz können wir einlesen und auf alle Daten anwenden, um den korrekt klassifizierten Anteil aller Datensätze zu bestimmen. Das folgende Programm gibt den Gesamtfehler und den genannten Anteil aus.
import numpy
import tensorflow.keras
network = tensorflow.keras.models.load_model('best.h5')
data = numpy.loadtxt('diabetes.csv', delimiter=',')
print(network.evaluate(data[:,:-1], data[:,-1]))
Es erzeugt die folgende Ausgabe.
# python diabetes_eval.py
...
[0.20267222449183464, 0.671875]
Die erste Zahl ist der Fehler (loss) und die zweite der Anteil korrekt klassifizierter Daten (acc), der also gut 67% beträgt.